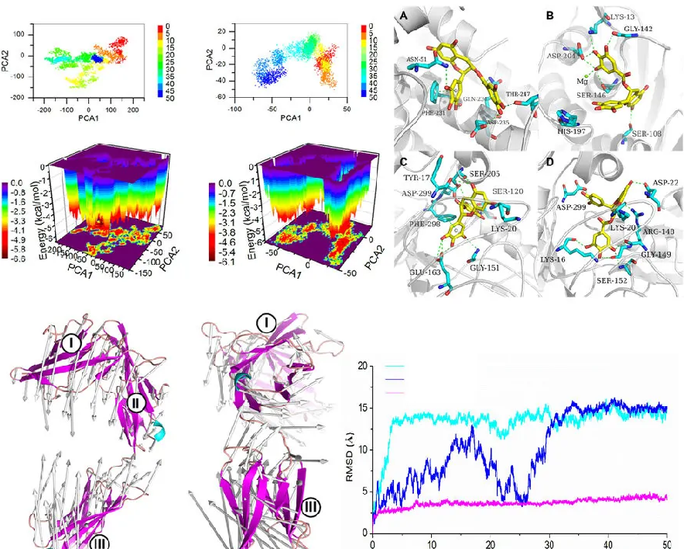
确定起始构型
进行分子动力学模拟的第一步是确定起始构型,一个能量较低的起始构型是进行分子模拟的基础,一般分子的起始构型主要来自实验数据或量子化学计算。 在确定起始构型之后要赋予构成分子的各个原子速度,这一速度是根据波尔兹曼分布随机生成的,由于速度的分布符合波尔兹曼统计,因此在这个阶段,体系的温度是恒定的。另外,在随机生成各个原子的运动速度之后须进行调整,使得体系总体在各个方向上的动量之和为零,即保证体系没有平动位移。
进入平衡相
由上一步确定的分子组建平衡相,在构建平衡相的时候会对构型、温度等参数加以监控。
进入生产相
进入生产相之后体系中的分子和分子中的原子开始根据初始速度运动,可以想象其间会发生吸引、排斥乃至碰
分子动力学
步骤
以下是做模拟的一般性步骤,具体的步骤和过程依赖于确定的系统或者是软件,但这不影响我们把它当成一个入门指南:
1)首先我们需要对我们所要模拟的系统做一个简单的评估, 三个问题是我们必须要明确的:
做什么(what to do)为什么做(why to do)怎么做(how to do)
2)选择合适的模拟工具,大前提是它能够实现你所感兴趣的目标,这需要你非常谨慎的查阅文献,看看别人用这个工具都做了些什么,有没有和你相关的,千万不要做到一半才发现原来这个工具根本就不能实现你所感兴趣的idea,切记! 考虑1:软件的选择,这通常和软件主流使用的力场有关,而软件本身就具体一定的偏向性,比如说,做蛋白体系,Gromacs,Amber,Namd均可;做DNA, RNA体系,首选肯定是Amber;做界面体系,Dl_POLY比较强大,另外做材料体系,Lammps会是一个不错的选择
考虑2:力场的选择。力场是来描述体系中最小单元间的相互作用的,是用量化等方法计算拟合后生成的经验式,有人会嫌它粗糙,但是它确确实实给我们模拟大系统提供了可能,只能说关注的切入点不同罢了。常见的有三类力场:全原子力场,联合力场,粗粒化力场;当然还有所谓第一代,第二代,第三代力场的说法,这里就不一一列举了。
再次提醒注意:必须选择适合于我们所关注体系和我们所感兴趣的性质及现象的力场。 3)通过实验数据或者是某些工具得到体系内的每一个分子的初始结构坐标文件,之后,我们需要按我们的想法把这些分子按照一定的规则或是随机的排列在一起,从而得到整个系统的初始结构,这也是我们模拟的输入文件。
4)结构输入文件得到了,我们还需要力场参数输入文件,也就是针对我们系统的力场文件,这通常由所选用的力场决定,比如键参数和非键参数等势能函数的输入参数。 分子动力学
5)体系的大小通常由你所选用的box大小决定,我们必须对可行性与合理性做出评估,从而确定体系的大小,这依赖于具体的体系,这里不细说了。6)由于初始构象可能会存在两个原子挨的太近的情况(称之为bad contact),所以需要在正式模拟开始的第一步进行体系能量最小化,比较常用的能量最小化有两种,最速下降法和共轭梯度法,最速下降法是快速移除体系内应力的好方法,但是接近能量极小点时收敛比较慢,而共轭梯度法在能量极小点附近收敛相对效率高一些,所有我们一般做能量最小化都是在最速下降法优化完之后再用共轭梯度法优化,这样做能有效的保证后续模拟的进行。 7)以平衡态模拟为例,你需要设置适当的模拟参数,并且保证这些参数设置和力场的产生相一致,举个简单的例子,gromos力场是用的范德华势双截断来定范德华参数的,若你也用gromos力场的话也应该用双截断来处理范德华相互作用。常见的模拟思路是,先在NVT下约束住你的溶质(剂)做限制性模拟,这是一个升温的过程,当温度达到你的设定后, 接着做NPT模拟,此过程将调整体系的压强进而使体系密度收敛。 经过一段时间的平衡模拟,在确定系统弛豫已经完全消除之后,就可以开始取数据了。如何判断体系达到平衡,这个问题是比较技术性的问题,简单的讲可以通过以下几种方式,一,看能量(势能,动能和总能)是否收敛;二,看系统的压强,密度等等是否收敛;三看系统的RMSD是否达到你能接受的范围,等等。
8)运行足够长时间的模拟以确定我们所感兴趣的现象或是性质能够被观测到,并且务必确保此现象出现的可重复性。
9)数据拿到手后,很容易通过一些可视化软件得到轨迹动画,但这并不能拿来发文章。真正的工作才刚刚开始——分析数据,你所感兴趣的现象或性质只是表面,隐含在它们之中的机理才是文章中的主题。